
整理 | 夕颜
出品 | CSDN(ID:CSDNnews)
人到中年,也许不到中年,不少人就已经被秃头问题困扰,贵为英国皇室,也没办法阻挡秃头这项“光荣的家族传统”。很多人认为,连皇家都治不了这种历史源远流长的疾病,看来那就不是差钱的事,秃头是真的难治!
秃头是难治,但科学医学发展日新月异,这不,秃头星人的福星来了!
腾讯 AI Lab联合团队首次破解秃头关键因素“SRD5A2”酶结构
11月17日,腾讯公布了一项人工智能助力药物发现的新进展。

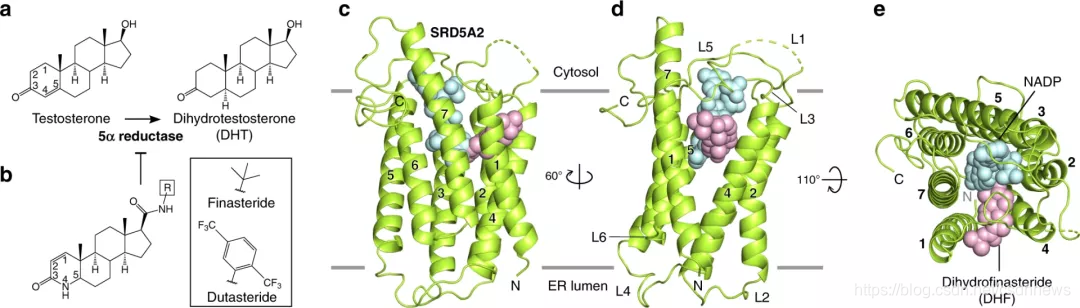
通过腾讯自研的提升蛋白质结构预测精度的新方法,联合研究团队首次解析了II型5a还原酶(SRD5A2)的三维结构,揭示了治疗脱发和前列腺增生的关键药物分子“非那雄胺”对于该酶的抑制机制,这将有助于深化研究相关疾病的病理学机制及药物优化。
此次,腾讯 AI Lab 采用“从头折叠”的蛋白质结构预测方法帮助解析了SRD5A2晶体结构,并通过自研AI工具“ tFold”有效提升了蛋白质结构预测精度,在科研突破中发挥了核心作用。除了在SRD5A2结构中的应用,这套方法还可以拓展应用于蛋白质分子和病理学机制的相关研究中。
登上 Nature 子刊
这项联合研究成果于近日登上了国际顶级期刊 Nature 子刊《 Nature Communications》。论文题为《人体类固醇II型5a还原酶与抗雄激素药物非那雄胺的结构研究》。
论文链接: https://www.nature.com/articles/s41467-020-19249-z
该论文由南科大生物系魏志毅副教授课题组与匹兹堡大学张诚教授、新加坡 A*STAR 研究所范昊研究员、腾讯 AI Lab 黄俊洲博士带领的研究小组合作完成。本次得到权威学术期刊发表及评审的高度评价,也验证了该成果对药物研发的创新价值。

为了更精准地预测SRD5A2 蛋白的三维结构,腾讯 AI Lab 科研团队采用了“从头折叠”(de novo folding)方法。
《Nature Communications》的一位评审对此创新方法给予了这样的评价:
作者能用预测的分子置换(MR)模型来确定晶体结构,这一点非常有趣。本评审认为该技术确实非常出色,整个X射线晶体学界将从该方法中受益匪浅。
很多人看到这些专业术语可能还是一头雾水,SRD5A2是什么东西?“从头折叠”是什么技术?AI工具“ tFold”又是怎么发挥作用的?为什么这些技术对于解决秃头问题意义重大?了解这些问题的答案,我们需要先了解一些导致秃头问题的病理常识。
人为什么会秃头?
首先,我们要知道,人为什么会秃头。
在人体内,性激素有促进性器官成熟、副性征发育及维持性功能等作用。
二氢睾酮是人体中已知最强的雄激素,对于人体的发育和生理活动至关重要,但同时也需要保持合理的平衡。一方面,二氢睾酮控制着男性性器官的发育,水平过低将导致男性性征缺陷。另一方面,水平过高又是导致前列腺增生和脱发的罪魁祸首。

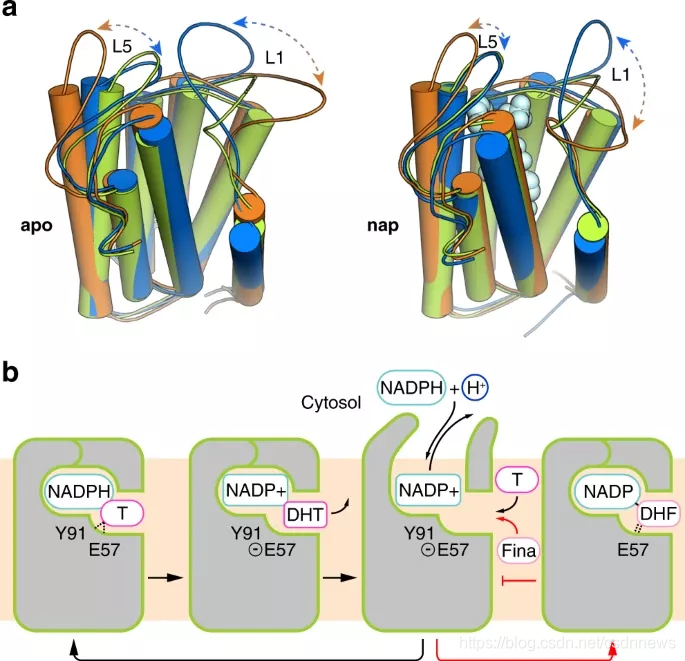
合成性激素依赖类固醇还原酶,二氢睾酮即由 SRD5A2 催化合成。因此,当患者因为二氢睾酮水平过高而出现前列腺增生和脱发问题时,可以通过抑制 SRD5A2 来降低患者二氢睾酮水平。作为SRD5A2 的高效抑制剂,非那雄胺(finasteride)被广泛用于治疗这类疾病。
然而现实中,尽管 SRD5A2 具有重要生理作用,但其高分辨率结构信息却十分缺乏,导致 SRD5A2 催化二氢睾酮合成的机理以及非那雄胺抑制 SRD5A2 酶活的机制并不清晰。
这是由于 SRD5A2 具有独特的七次跨膜结构,其与人类全部已知结构的蛋白在结构上存在较大差异,难以通过“模板建模”(template-based modeling)方法获得初始构型来解析晶体数据。同时,又因为 SRD5A2 是一类多次跨膜蛋白,使得传统的用于获取蛋白质晶体相位信息的“重原子替代”(Heavy-atom derivatization)方法亦难以奏效。
“从头折叠”新方法破解晶体学难题
到这里,我们基本上可以搞清楚了,为解决秃头问题,找到一个简单的方法为SRD5A2“建模”,搞清楚它的结构信息是非常关键的一步。
为了解决这一难题,腾讯 AI Lab 科研团队采用了难度更高的“从头折叠”(de novo folding)方法来预测 SRD5A2 蛋白的三维结构,并将其用于“分子置换”(molecular replacement, MR)的初始构型来解析晶体数据。
所谓“从头折叠”,是相对于“模板建模”的一种蛋白质结构预测方法。“模板建模”是目前最普遍的蛋白结构预测手段,但有一个使用前提——人类已知的蛋白结构数据库(即PDB)当中,必须存在和预测的蛋白相似的结构,否则就无法使用。而腾讯AI Lab采用的“从头折叠”方法则跳出了这个限制,可以不依赖于模板来预测蛋白结构。
但是在这里又存在一个问题:此前,通过“从头折叠”方法预测的蛋白质结构精度不高,难以满足晶体数据解析的精度需要。
为了获得更高精度的“从头折叠”的结构模型,腾讯采用了自研AI工具——tFold 工具,为分子置换方法提供相位,继而解析确定2.8Å 原子级别精度的SRD5A2晶体结构。
这一结果能直接推进我们对体内 SRD5A2 活性失调引发的各类疾病的理解,进而为基于 SRD5A2 结构的药物开发提供更多有价值的参考信息。
自研冠军级 tFold 工具突破蛋白质结构预测精度
腾讯 AI Lab 自研的 tFold 工具正是破解 SRD5A2 蛋白结构这一重要难题的关键。
为了提升“从头折叠”方法(又称“自由建模”)的精度,tFold 工具通过三项技术创新,实现了蛋白结构预测精度的大幅提升。
多数据来源融合
首先,实验室研发了“多数据来源融合”(multi-source fusion)技术,来挖掘多组多序列联配(multiplesequence alignment, MSA)中的共进化信息。因为蛋白序列的数据库有多个,在不同的数据库中,采用不同的检索方法以及不同的参数下,相同的输入序列可能对应不同的MSA,但这些MSA都包含有意义的共进化信息,因此需要通过一定手段将其合并所以,使用此手段可以实现这样的效果。
深度交叉注意力残差网络
然后,借助 “深度交叉注意力残差网络” (deep cross-attention residual network,DCARN),这是一种面向强噪声或者高度冗余数据的特征学习方法,其核心在于利用互相交叉的2D注意力机制,来更有效的捕捉远程的残基对相互作用。这种2D注意力机制的表现能力,比通常的深度卷积神经网络更强,因此能极大提高一些重要的蛋白2D结构信息(如:残基对距离与取向矩阵)的预测精度。
模板辅助自由建模
最后,通过一种新颖的“模板辅助自由建模“(Template-based Free Modeling, TBFM)方法,将自由建模(Free Modeling, FM)和模板建模(Template-based Modeling, TBM)生成的3D模型中的结构信息加以有效融合,从而大大提高了最终3D建模的准确性。
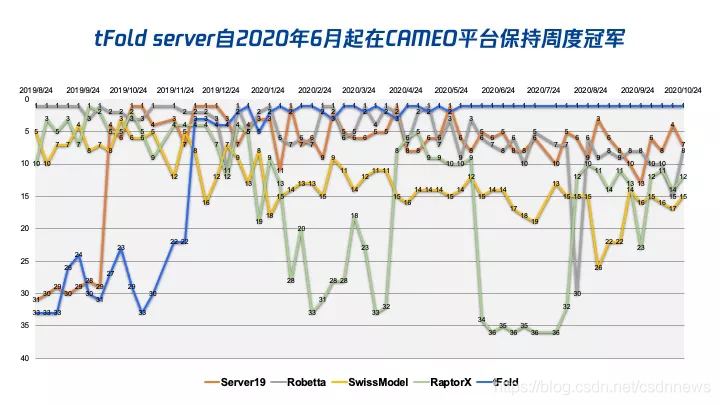
在研究方面,tFold 平台已在国际公认最权威的测试平台CAMEO上显露出价值。腾讯 AI Lab 于2020年初在CAMEO平台注册了自动化蛋白结构预测服务器 tFold server,并自2020年6月起至今一直保持周度(图1)、月度、季度、半年度冠军。
在应用方面,tFold server的公测版也已经在腾讯「云深智药」平台发布。
官网链接: https://drug.ai.tencent.com/console/cn/tfold
在这个平台上,用户可以手动输入待预测的氨基酸序列或从本地上传FASTA 格式的序列文件。在经过一定时间的计算之后,用户即可得到使用“从头折叠”方法预测得到高精确度蛋白结构(下图)。
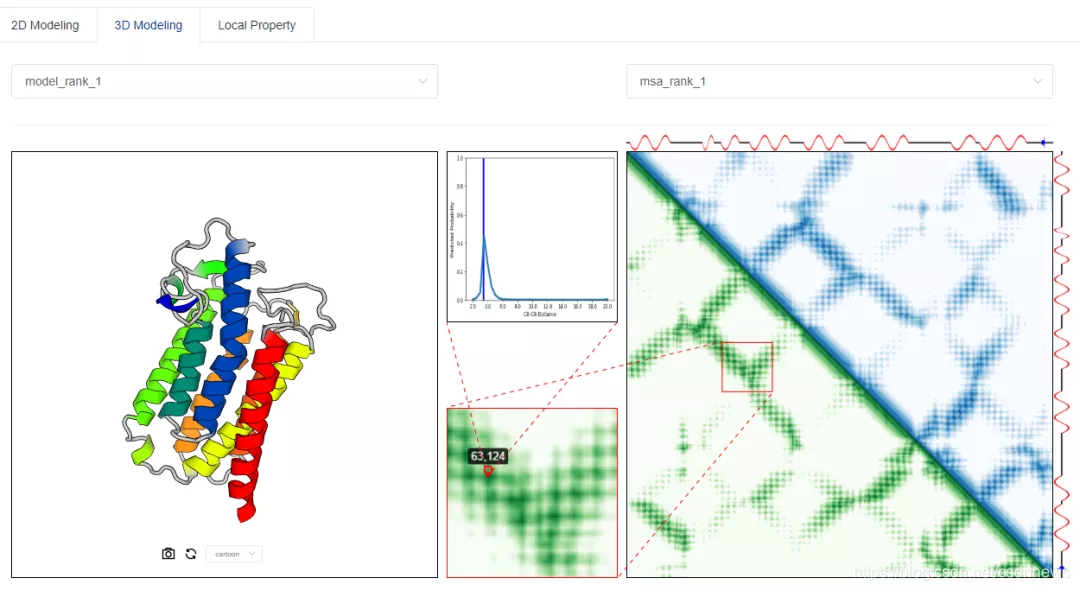
tFold server的3D Modeling输出页面。左边部分为从头折叠得到的3D蛋白模型;右边部分是该3D模型在给定的预测残基对距离矩阵下的偏差。
医疗AI大浪淘沙,但蓝海依然辽阔
本论文中解决秃头问题的关键工具tFold所依托的「云深智药」平台,是腾讯发布的首个AI驱动的药物发现平台,整合了腾讯 AI Lab和腾讯云在前沿算法、优化数据库以及计算资源上的优势,初衷是帮助用户大幅度减少寻找潜在活性化合物的时间和成本。
目前,「云深智药」的五大模块已覆盖临床前新药发现全流程,包括蛋白质结构预测、分子生成模块、虚拟筛选、ADMET预测和合成路线规划模块。
据了解,该平台的逆合成算法也已取得了一定进展,计划于明年上线,以及其它小分子和大分子药物发现功能模块也将逐步上线。
除药物研发以外,腾讯 AI Lab 也在影像筛查、病理诊断、病理研究等多个医疗领域持续探索,也有了一些成熟的应用实践,比如中国首款智能显微镜,宫颈液基细胞(TCT)标本临床样本观察、筛选、标记功能,能够准确区分神经胶质瘤(大脑最常见肿瘤)的不同亚型并进行分级的算法,等等,都是腾讯 AI Lab的应用成果。
随着2018年刮起的医疗AI热,像腾讯「云深智药」这样的医疗AI平台还有很多,有的背靠大型科技企业,有着充足的研发资金与技术支持,有的术业有专攻,企业小而精,专注于技术的落地与应用。
2019年,大浪淘沙,医疗AI的融资额总体呈降低趋势,医疗AI热暂缓,但头部公司的融资额在增加,留下的是实力派。据IDC统计数据,到2025年人工智能应用市场总值将达到1270亿美元,其中医疗行业将占市场规模的五分之一。如今,医疗AI市场也和众多AI领域一样冷静下来,进入沉淀期,这对药物研发和病理研究领域来说是件好事,因为AI在这片领域还大有可为,尤其经过此次新冠疫情,AI在医学领域的价值更是得到了实践的验证。
谁能在这片蓝海中抢下一城,成为医疗AI领域的“霸主”,还要凭技术研究实力,以及最终的实践应用能力说话,我们且静观其变。
